Abstrak
Genus Tetramesa Walker (Hymenoptera: Eurytomidae) mencakup lebih dari 200 spesies tawon herbivora yang memakan rumput secara eksklusif. Survei lapangan terbaru di Afrika Selatan untuk program pengendalian hayati rumput telah mengungkap keragaman besar potensi Tetramesa pada rumput Afrika. Di sini, urutan genetik mitokondria (sitokrom c oksidase I [COI]) dan nuklir (28S) digunakan untuk membandingkan keluaran dari tujuh metode delimitasi spesies yang populer dan untuk memandu pembuatan batas spesies konsensus untuk taksa Tetramesa putatif dan kerabat dekatnya. Selain itu, wilayah nuklir digunakan untuk menjalankan analisis tanggal yang menerapkan laju jam molekuler. Hasil delimitasi spesies konsensus menemukan 35 unit taksonomi operasional molekuler (MOTU) dalam data COI dan 21 MOTU dalam data 28S. Dari 35 MOTU COI, terdapat 17 taksa Tetramesa putatif (16 taksa Afrika selatan yang baru dan 1 spesies Belahan Bumi Utara yang telah dideskripsikan, Tetramesa romana ), 13 di antaranya menunjukkan bukti spesialisasi pada satu tanaman inang. Sebagai perbandingan, dari dua puluh satu MOTU 28S, terdapat 5 taksa Tetramesa putatif (4 taksa Afrika selatan yang baru dan 1 T. romana ), yang semuanya menunjukkan bukti spesifisitas inang. Analisis yang diberi tanggal menunjukkan bahwa genus Tetramesa berasal sekitar ~67,1 juta tahun lalu. Terdapat bukti diversifikasi cepat dalam klade Belahan Bumi Selatan antara 5 dan 15 juta tahun lalu, yang bertepatan dengan perluasan padang rumput dan fluktuasi iklim di Afrika pada saat itu yang mungkin telah mendorong spesialisasi inang. Hasil saat ini memberikan wawasan berharga ke dalam keanekaragaman dan pola evolusi skala yang lebih luas dalam kelompok mikrohimenoptera Belahan Bumi Selatan ini.
PERKENALAN
Genus Tetramesa Walker (Hymenoptera: Eurytomidae) adalah kelompok tawon herbivora yang memakan rumput, biasanya hanya berspesialisasi pada satu spesies atau genus atau pada genus rumput yang terkait erat (Claridge 1961 ; Phillips 1936 ). Kelompok tawon ini menarik bagi praktisi pengendalian hayati karena penggunaannya dalam program yang menargetkan rumput invasif (Sutton et al. 2019 , 2021 ; Witt & McConnachie 2004 ), terutama setelah keberhasilan Tetramesa romana Walker untuk pengendalian Arundo donax L. di sepanjang Rio Grande di Texas (AS) dan di Meksiko (Goolsby & Moran 2009 ; Moran & Goolsby 2009 ; Racelis et al. 2010 ).
Hampir semua upaya pengambilan sampel dalam mengumpulkan dan mendeskripsikan spesies Tetramesa telah dilakukan di Belahan Bumi Utara (NH) (Al-Barrak 2006 ). Hanya empat spesies Afrika yang dideskripsikan yaitu Tetramesa aristidae Risbec dari Senegal, Tetramesa decaryi Risbec dan Tetramesa tananarivense Risbec dari Madagaskar, dan Tetramesa macalusoi De Stefani dari Somalia (van Noort 2020 ). Survei terbaru oleh Sutton et al. ( 2021 ) dan Yell et al. ( 2024 ) pada rumput asli Afrika dan analisis filogenetik selanjutnya (Sutton et al. 2023 ; van Steenderen et al. 2023 ) telah mengungkapkan serangkaian taksa Tetramesa baru , terutama pada spesies inang Eragrostis Wolf dan Sporobolus R.Br.
Selain sejarah taksonomi Tetramesa yang berbelit-belit ( Claridge 1961 ; Gahan 1924 ; Phillips & Emery 1919 ) dan kesamaan morfologi antara dewasa dan larva di berbagai spesies, terdapat banyak kesenjangan pengetahuan dalam kelompok ini. Ini termasuk yang berikut: (1) keanekaragaman spesies pada rumput asli Afrika, yang sangat penting karena Afrika merupakan donor utama rumput invasif di seluruh dunia (Visser et al. 2016 ); (2) pemahaman tentang spesifisitas inang dan mekanisme reproduksi dan perilaku dalam genus; (3) keahlian taksonomi dalam identifikasi morfologi; (4) pola radiasi historis menggunakan metode penanggalan molekuler; (5) filogeografi umum kelompok; dan (6) menentukan batas spesies molekuler spesifik penanda.
Metode delimitasi spesies molekuler menggunakan data genetik untuk memperkirakan batas spesies dan jumlah kelompok taksonomi berbeda yang dihasilkan dalam sistem studi tertentu. Metode ini bergantung pada konstruksi pohon filogenetik atau matriks jarak genetik (Fontaneto et al. 2007 ). Alat genetik seperti ini telah menjadi pelengkap yang berharga untuk taksonomi morfologi tradisional, terutama untuk taksa yang terdiri dari kelompok yang secara morfologis tidak dapat dibedakan dan mungkin mengandung beberapa spesies kriptik (Anguita-Salinas et al. 2017 ), secara morfologis plastis dalam menanggapi lingkungannya, yang mengarah pada konvergensi atau divergensi morfologis (Guo et al. 2018 ), berukuran mikroskopis (Mills et al. 2017 ), atau menjalani tahap larva yang menantang atau tidak mungkin diidentifikasi (Collin et al. 2020 ).
Kolaborasi telah dimulai antara Afrika Selatan dan Australia di mana para peneliti tengah mencari agen pengendali hayati potensial yang dapat digunakan untuk menargetkan rumput Afrika invasif di Australia, yaitu Eragrostis curvula (Schrad.) Nees (Rumput cinta Afrika), Andropogon gayanus Kunth (Rumput Gamba), Sporobolus natalensis (Steud.) T. Durand & Schinz dan Sporobolus pyramidalis (Steud.) T. Durand & Schinz (Rumput ekor tikus raksasa). Saat ini juga sedang dilakukan survei untuk mencari agen prospektif untuk Megathyrsus maximus (Jacq.) BKSimon & SWLJacobs (Rumput Guinea) bekerja sama dengan para peneliti dari Amerika Serikat. Survei jangkauan asli pada rumput Afrika di Afrika Selatan sejak 2017 telah menghasilkan sejumlah spesimen Tetramesa yang tidak teridentifikasi , tetapi kurangnya spesies Tetramesa yang dideskripsikan di Afrika telah membuat pengujian spesifisitas inang berbasis lapangan menjadi sulit dilakukan (Sutton et al. 2019 , 2021 ; van Steenderen et al. 2023 ). Kurangnya keahlian taksonomi juga telah membatasi pengembangan taksa Tetramesa sebagai agen pengendalian hayati yang potensial, karena identifikasi yang benar dan asosiasi tanaman inang sangat penting untuk menilai spesifisitas inang.
Penelitian ini bertujuan untuk membedakan secara genetik antara kelompok Tetramesa Afrika , menentukan spesifisitas inangnya, dan memperoleh pemahaman yang lebih baik tentang proses diversifikasi di antara taksa yang memakan rumput inang yang berbeda. Di sini, kami menggunakan COI mitokondria dan wilayah gen 28S rDNA nuklir untuk memperkirakan batas spesies Tetramesa dan juga menyelidiki waktu divergensi potensial menggunakan laju jam molekuler. Memahami hubungan dan sejarah filogenetik kelompok tawon ini berharga tidak hanya untuk penelitian pengendalian biologis tetapi juga untuk keanekaragaman hayati dan konservasi invertebrata Afrika yang lebih luas.
BAHAN DAN METODE
Protokol laboratorium
Urutan sitokrom c oksidase I (COI) mitokondria diperoleh menggunakan primer forward FWPTF1 (5′-CCTGGTTCTTTRATTGGTAATGATC-3′) (Li et al. 2010 ) dan reverse LepR1 (5′-TAAACTTCTGGATGTCCAAAAA-3′) (Hebert et al. 2004 ). Setiap reaksi berantai polimerase (PCR) dilakukan dalam volume 25-μL, yang terdiri dari campuran master PCR 12,5-μL (2× iTaq™ Universal SYBR® Green Supermix, Bio-Rad, Nomor Katalog 1725121), primer forward dan reverse 2-μL (konsentrasi 10-μM) dan DNA cetakan (∼5–10 ng/μL). Kondisi siklus termal adalah sebagai berikut: denaturasi 5 menit pada 94°C, diikuti oleh 38–40 siklus 40 detik pada 94°C; 35 detik pada 53°C; 45 detik pada 72°C, dan ekstensi akhir 10 menit pada 72°C. Urutan 28S rRNA nuklir diperoleh dengan menggunakan primer D2F (5′-AGTCGTGTTGCTTGATAGTGCAG-3′) dan D2R (5′-TTGGTCCGTGTTTCAAGACGGG-3′) dalam Campbell et al. ( 1994 ), di mana kondisi siklus termal adalah sebagai berikut: denaturasi 5 menit pada 94°C, diikuti oleh 35 siklus 1 menit pada 94°C; 1 menit pada 55°C; 1,5 menit pada suhu 72°C, dan perpanjangan akhir selama 10 menit pada suhu 72°C (dimodifikasi dari Schmidt et al. 2006 ). Spesimen dibagi menjadi dua morfotipe utama, yaitu yang memiliki dan tidak memiliki bercak pronotal (masing-masing PNS dan NPNS) (Gambar 1 ).

Bioinformatika
Pengurutan dan penyelarasan nukleotida
Produk PCR yang berhasil diperkuat dikirim ke Macrogen, Inc. ( https://dna.macrogen-europe.com/eng/ ) di Belanda untuk pemurnian dan pengurutan pasca-PCR. Wilayah 28S diurutkan dalam arah maju dan wilayah COI dalam arah sebaliknya untuk menghindari wilayah poli-T di ujung 5′ wilayah kode batang. Kualitas setiap kromatogram DNA diperiksa secara manual menggunakan Chromas v2.6.4 (Technelysium Pty Ltd) dan dipangkas dengan tepat. Urutan diselaraskan secara daring menggunakan parameter default di MAFFT v7 ( https://mafft.cbrc.jp/alignment/server/ ) (Katoh et al. 2019 ) dan ditingkatkan secara manual jika sesuai. Penyelarasan COI diterjemahkan menjadi asam amino dalam kerangka baca terbuka (ORF) terpanjangnya untuk menghilangkan kodon stop dan indel (Buhay 2009 ).
Analisis filogenetik
Urutan 28S dan COI tambahan diperoleh dari basis data GenBank ( https://www.ncbi.nlm.nih.gov/genbank/ ) dan BOLD ( https://www.boldsystems.org/index.php/ ) dan tercantum dalam Tabel S1 dan S2 . Kelompok luar dipilih berdasarkan karya Chen et al. ( 2004 ). Wilayah gen COI mitokondria dan 28S nuklir dianalisis secara terpisah karena adanya klade yang belum terpecahkan dalam kedua filogeni. Model substitusi optimal dan filogeni kemungkinan maksimum (ML) diperkirakan menggunakan server daring IQ-TREE ( http://iqtree.cibiv.univie.ac.at/ ) (Chernomor et al. 2016 ; Nguyen et al. 2015 ; Trifinopoulos et al. 2016 ), di mana model terbaik ditemukan oleh ModelFinder (Kalyaanamoorthy et al. 2017 ). Parameter default diterapkan dalam IQ-TREE, dengan 1000 pengulangan bootstrap yang sangat cepat.
Set data COI dipartisi berdasarkan posisi kodon, di mana model optimalnya adalah TIM3 + F + Γ (Posisi 1) dan TIM + F + Γ (Posisi 2 dan 3) menurut kriteria informasi Bayesian (BIC). Model optimal untuk set data 28S adalah GTR + F + I + Γ. Filogeni inferensi Bayesian (BI) dibuat dalam MrBayes v3.2.6 (Huelsenbeck & Ronquist 2001 ), menggunakan pohon awal acak, 4 rantai (3 panas dan 1 dingin), 2 kali uji coba, 25 juta generasi, dan parameter model yang diperoleh dari IQ-TREE. Pohon diambil sampelnya setiap 1000 generasi dengan burn-in 25%. Tracer v1.7 (Rambaut et al. 2018 ) digunakan untuk memeriksa konvergensi Markov chain Monte Carlo (MCMC) (skor ukuran sampel efektif [ESS] >200). Nilai probabilitas posterior antara 0,9 dan 1,0 dianggap sangat didukung. Semua analisis dilakukan pada server daring CIPRES Science Gateway v3.3 ( http://www.phylo.org/ ) (Miller et al. 2010 ). File pohon keluaran disiapkan menggunakan paket R ‘ggtree’ (Yu et al. 2017 ) dan ‘treeio’ (Wang, Lam, et al. 2020 ) dalam R v4.0.2 (Tim Inti R 2023 ).
Konvensi penamaan
Klade diberi nama sebagai berikut: MORPHOTYPE.HOSTPLANT.NUMBER di mana morfotipe merujuk pada keberadaan (PNS) atau ketiadaan (NPNS) bercak pronotal atau tidak diketahui (U). Tanaman inang disingkat menjadi huruf pertama genus, diikuti oleh beberapa karakter yang dapat dikenali yang menunjukkan nama spesies. Misalnya, PNS.ECUR.1 berarti Nomor Klade 1, yang mewakili morfotipe bercak pronotal (PNS), yang dikumpulkan pada E. curvula (ECUR). Dalam beberapa kasus, tanaman inang tidak diketahui (U), atau klade mungkin terdiri dari beberapa tanaman inang (MIX). Nomor klade filogenetik yang diurutkan disediakan untuk navigasi cepat, dan nomor klade konsensus didasarkan pada keluaran pembatasan spesies.
Pembatasan spesies
Karena metode delimitasi spesies cenderung menghasilkan keluaran yang bervariasi (Blair & Bryson 2017 ; Hofmann et al. 2019 ; Ranasinghe et al. 2022 ; van Steenderen & Sutton 2022 ), tujuh metode independen dieksplorasi untuk memperkirakan batas spesies (yaitu, jumlah unit taksonomi operasional molekuler [MOTU]) dalam set data genetik Tetramesa COI dan 28S. Metode tersebut adalah (1) Automatic Barcode Gap Discovery (ABGD), (2) Assemble Species by Automatic Partitioning (ASAP), (3) Bayesian Poisson Tree Processes (bPTP), (4) Poisson Tree Processes (PTP), (5) Multi-rate Poisson Tree Processes (mPTP), (6) Generalised Mixed Yule Coalescent (GMYC) dan (7) Haplowebs. Outgroup dikecualikan dari semua analisis. Nilai divergensi intraspesifik dan interspesifik untuk klade terpilih dihitung dalam MEGA11 (Kumar et al. 2018 ) menggunakan jarak yang tidak dikoreksi. Kolom konsensus disusun berdasarkan tren yang ditemukan di semua metode dan asosiasi tanaman inang yang diamati di lapangan dan laboratorium. Singleton (MOTU dengan hanya satu urutan representatif) tidak disertakan dalam delimitasi konsensus. Lihat Data S1 untuk detail metodologi delimitasi spesies lebih lanjut.
Penanggalan jam molekuler
Beberapa fosil chalcidoid tertua (Eulophidae dan Trichogrammatidae) ditemukan terawetkan dalam amber di Ethiopia dan diperkirakan berasal dari pertengahan Cretaceous ~93–95 juta tahun lalu (Schmidt et al. 2010 ). Fosil eurytomid yang tercatat hanya dideskripsikan sebagai Eurytoma sepulta Brues (Miosen), Eurytoma sequax Brues (Miosen) dan Decatoma antiqua Scudder (Oligosen) (Yoshimoto 1975 ). Tanggal-tanggal ini tidak digunakan sebagai titik kalibrasi dalam studi ini karena Eurytoma bukan merupakan kelompok monofiletik (Chen et al. 2004 ).
Karena tidak adanya fosil yang dapat diandalkan atau titik kalibrasi geologi lainnya yang berhubungan dengan Eurytomidae, laju jam nuklir sebesar 0,06 ± 0,03% per juta tahun (Papadopoulou et al. 2010 ) diterapkan pada wilayah 28S rDNA untuk memperkirakan waktu divergensi. Wilayah COI nuklir daripada wilayah mitokondria dipilih untuk analisis penanggalan karena sifat konservatif RNA nuklir dan keandalannya yang diketahui dalam menyimpulkan hubungan filogenetik yang dalam (Hillis & Dixon 1991 ; Hwang & Kim 1999 ). Selain itu, Chen et al. ( 2004 ) merekomendasikan penggunaan wilayah nuklir daripada wilayah mitokondria untuk penyelidikan lebih lanjut tentang Eurytomidae karena sebagian dinamika pewarisan dan proses evolusi DNA mitokondria yang tidak diketahui dalam kelompok hymenopteran ini.
Dua analisis Bayesian independen dijalankan dalam BEAST v2.6.3 (Suchard et al. 2018 ), menerapkan jam molekuler yang rileks (lognormal tidak berkorelasi) (opsi mode laju jam yang ditetapkan otomatis tidak dipilih dalam BEAST). Model evolusi yang sama diterapkan untuk wilayah 28S (Data S1 ) (GTR + F + I + Γ). Prior pohon ditetapkan ke model Yule, dan laju jam sebelumnya ditetapkan ke distribusi lognormal yang berpusat pada 0,0003, dengan deviasi standar 0,00015. Opsi default diterapkan untuk pengaturan operator dan prior yang tersisa. Setiap analisis dijalankan selama 50 juta generasi, dengan pohon diambil sampelnya setiap 1000. Pohon kredibilitas klade maksimum menggunakan tinggi median dibangun dengan TreeAnnotator v1.8.3 (Suchard et al. 2018 ), di mana burn-in pohon ditetapkan menjadi 75%. File log dilihat di Tracer v1.7 (Rambaut et al. 2018 ) untuk memeriksa konvergensi dan skor ESS >200. File pohon BEAST dibaca ke dalam R melalui fungsi phyloch::read.beast() (Heibl 2019 ), dan filogeni yang diberi tanggal diplot menggunakan fungsi strap::geoscalePhylo() (Bell & Lloyd 2015 ). Plot garis keturunan melalui waktu (ltt) dibuat menggunakan fungsi ape::ltt.plot() (Paradis et al. 2004 ).
Gambar 5 memuat informasi yang mengindikasikan (i) estimasi asal usul ordo Hymenoptera dan Panorpoid kelompok batang (Grimaldi & Engel 2005 ), (ii) estimasi asal usul dan radiasi angiospermae berdasarkan data molekuler (Li et al. 2019 ), (iii) catatan fosil hymenoptera (famili Xyelidae) tertua yang diketahui yang ditemukan di Australia (Riek 1955 ; Yoshimoto 1975 ), (iv) kemungkinan asal usul Chalcidoidea (Heraty & Darling 2009 ), (v) usulan asal usul dan radiasi angiospermae berdasarkan data fosil (Li et al. 2019 ), tetapi (vi) kemungkinan kelompok batang mungkin berasal dari Trias Tengah berdasarkan penemuan serbuk sari mirip angiospermae, (vii) beberapa fosil chalcidoid paling awal yang ditemukan di Ethiopia (Schmidt et al. 2010 ), (viii) periode radiasi chalcidoid (Heraty & Darling 2009 ), (ix) Perluasan padang rumput Miosen di Afrika (Jacobs 2004 ; Uno et al. 2016 ) dan (x) perkiraan tanggal dua fosil Eurytoma dan satu fosil Decatoma (Yoshimoto 1975 ).
HASIL
Pembatasan spesies
Wilayah COI berisi 35 konsensus MOTU, yang terdiri dari 17 taksa Tetramesa putatif (Gambar 2 ; Konsensus Klade 1–13 dan 24–27). Dari jumlah tersebut, ada sembilan MOTU Tetramesa potensial spesifik inang dalam kelompok PNS (Gambar 2 ; Konsensus Klade 1–3, 5, 6 dan 8–11). Taksa ‘spesifik inang’ merujuk pada taksa yang dibatasi untuk memakan hanya satu spesies inang rumput. Tingkat spesifisitas ini sering kali diperlukan untuk program pengendalian biologis. Selain itu, empat MOTU NPNS bersifat spesifik inang dan kemungkinan termasuk dalam genus Tetramesa karena pengelompokannya yang dekat dengan T. romana dan Tetramesa bambusae yang teridentifikasi secara positif (Gambar 2 ; Konsensus Klade 24–27). Sebagai perbandingan, data 28S berisi 21 MOTU konsensus, yang terdiri dari 5 taksa Tetramesa spesifik inang PNS (Gambar 4 ; Klade Konsensus 1, 2 dan 4–6). Empat dari lima taksa ini mewakili taksa Tetramesa Afrika Selatan yang baru , sedangkan takson yang tersisa adalah spesies NH yang dideskripsikan, T. romana .

Data COI
Jumlah MOTU dan singleton yang diestimasikan berkisar antara 40 hingga 87 dan dari 11 hingga 54, masing-masing, di seluruh metode delimitasi. Jumlah MOTU untuk setiap metode delimitasi adalah: ABGD = 53, ASAP = 87, bPTP = 87, PTP = 73, mPTP = 40 dan GMYC = 71 (klaster = 42, entitas = 71, interval kepercayaan [CI] klaster = 36–43 dan entitas CI = 62–77) (Gambar 2 dan 3 ). Metode mPTP adalah yang paling konservatif dan memperkirakan jumlah MOTU dan singleton paling sedikit dibandingkan dengan metode lainnya, sedangkan metode ASAP dan bPTP memperkirakan jumlah MOTU terbesar (Gambar 3 ).
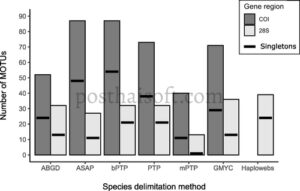
Hasil delimitasi di antara keenam metode tersebut secara umum kongruen, tetapi keberadaan singleton dan polytomies mengakibatkan beberapa variasi dalam estimasi MOTU (Gambar 2 ). Ada 13 konsensus MOTU di bagian PNS dari filogeni (Konsensus Clades 1–13) dan 8 di polytomi NPNS di bawahnya (Konsensus Clades 14–21; Gambar 2 ). Metode GMYC adalah satu-satunya yang mengelompokkan tawon PNS Eragrostis biflora (PNS.EBI.1, Clade 2) dengan kelompok PNS E. curvula (PNS.ECUR.1, Clade 1) sebagai satu MOTU unik, sedangkan metode mPTP yang lebih konservatif mengelompokkan Clades 1–6 sebagai MOTU tunggal (Gambar 2 ). Karena divergensi sekuens sebesar 2,5%, dan pemanfaatan spesies inang yang berbeda, ini diperlakukan sebagai dua MOTU terpisah.
Perpecahan dalam Klade 10 yang ditemukan oleh metode GMYC lebih mungkin merupakan perbedaan tingkat populasi karena pemisahan geografis antara spesimen KwaZulu-Natal dan Eastern Cape daripada perbedaan tingkat spesies. Tawon yang dikumpulkan pada Sporobolus africanus (PNS.SAFR.1, Klade 11) didukung sebagai MOTU unik oleh semua metode kecuali mPTP. Demikian pula, klade PNS.MIXED.1 (Klade 12) dan PNS.MIXED.2 (Klade 13) didukung sebagai MOTU terpisah, kecuali oleh ABGD untuk PNS.MIXED.2, yang membagi dua sampel menjadi sampel tunggal.
Sungguh mengejutkan bahwa dua spesimen yang dikumpulkan dari E. curvula (LMY 2021 00282 dan LMY 2021 00289) termasuk dalam klade NPNS S. pyramidalis . Namun, keduanya didukung sebagai MOTU terpisah oleh metode delimitasi ASAP, PTP, dan GMYC.
Klade NPNS.HHIR.1 (Klade 18) didukung sebagai MOTU tunggal di semua metode kecuali ABGD, yang mengecualikan satu sampel sebagai singleton. Klade NPNS.U.1 dan NPNS.U.2 secara tentatif diperlakukan sebagai satu kelompok karena divergensi sekuens yang rendah di antara keduanya (1,8%). Hanya satu sekuens dari Bavaria, Jerman, yang dikelompokkan dengan spesimen Kanada ini. Semua spesimen Jerman lainnya termasuk dalam klade NPNS.U.4 (Klade 28) dan NPNS.U.5 (Klade 29). Klade 19, 20, 28 dan 29 bersumber dari basis data BOLD, yang mana mereka ditugaskan ke genus Tetramesa berdasarkan Nomor Indeks Kode Batang (BIN), bukan pada identifikasi morfologi. Hasil saat ini menunjukkan bahwa spesimen ini lebih mungkin termasuk ke dalam genus eurytomid yang berkerabat dekat dan bukan ke Tetramesa .
Sampel dalam NPNS.MIXED.3 (Clade 21) didukung dengan baik sebagai MOTU tunggal, dan karena tujuh sampel Bruchophagus membentuk kelompok saudara dengannya, sampel-sampel ini kemungkinan juga termasuk dalam genus Bruchophagus . Meskipun hanya metode ABGD, mPTP dan GMYC yang mendukung NPNS.AGAYA.1 (Clade 23) sebagai MOTU tunggal, sampel ini diperlakukan demikian karena rumput inang unik ( A. gayanus ) tempat spesimen dikumpulkan di Zimbabwe. Akan tetapi, diperlukan lebih banyak sekuens untuk membuat kesimpulan lebih lanjut mengenai kelompok potensial ini.
Meskipun klade NPNS.HHIR.2 (Klade 22), NPNS.HHIR.3 (Klade 24), PNS.ADON.1 (Klade 25), NPNS.U.3 (Klade 26), NPNS.ETEF.1 (Klade 27), NPNS.U.4 (Klade 28), NPNS.U.5 (Klade 29) dan NPNS.ERIG.1 (Klade 30) belum terpecahkan dalam filogeni (yaitu, politomi), klade-klade tersebut secara umum didukung dengan baik oleh semua metode pembatasan (Gambar 4 ). Tidak ada metode pembatasan yang menyertakan T. bambusae dengan klade T. romana (PNS.ADON.1, Klade 25).

data 28 detik
Jumlah MOTU dan singleton yang diperkirakan berkisar antara 13 hingga 43 dan dari 1 hingga 24, masing-masing, di seluruh metode delimitasi. Jumlah MOTU untuk setiap metode delimitasi adalah: ABGD = 29, ASAP = 27, bPTP = 36, PTP = 36, mPTP = 13, GMYC = 36 (klaster = 23, entitas = 36, klaster CI = 15–28 dan entitas CI = 20–43) dan Haploweb = 43 (Gambar 3 dan 4 ). Seperti pada data COI, metode mPTP adalah yang paling konservatif. Meskipun metode yang tersisa memperkirakan jumlah MOTU yang sama, Haploweb dan GMYC menghasilkan perkiraan tertinggi (Gambar 3 ).
Tujuh metode delimitasi pada umumnya kongruen (Gambar 4 ). Klade konsensus 1, 2, 12 dan 14 dipertahankan sebagai MOTU unik karena pemanfaatan genus rumput inang yang berbeda (yaitu, Eragrostis spp., Sporobolus spp. dan Hyparrhenia hirta ).
Ada lima MOTU konsensus dalam kelompok yang dianggap termasuk dalam genus Tetramesa (Gambar 4 ). Ini termasuk tawon yang dikumpulkan pada Eragrostis rigidior (PNS.ERIG.1; Konsensus Klade 1), S. pyramidalis + Sporobolus sp. (PNS.SAFR.1; Konsensus Klade 2), Konsensus Klade 3 yang terdiri dari spesimen PNS yang belum terpecahkan, T. romana (PNS.ADON.1) + T. bambusae (Konsensus Klade 4), H. hirta (NPNS.HHIR.1; Konsensus Klade 5) dan A. gayanus (NPNS.AGAYA.1; Konsensus Klade 6). Meskipun belum terselesaikan, dua spesimen NPNS.AGAYA.1 (Konsensus Klade 6) kemungkinan merupakan kelompok saudara tunggal bagi NPNS.HHIR.1 (Konsensus Klade 5), dan karena mereka dikumpulkan pada inang yang berbeda, A. gayanus , mereka telah diperlakukan sebagai MOTU putatif tunggal.
Spesimen GenBank yang direkam sebagai Tetramesa , AY599281 dan DQ080114, didukung sebagai MOTU unik hanya oleh mPTP dan sebagai singleton oleh lima dari enam metode yang tersisa (Gambar 4 ). Karena penempatannya dalam filogeni, spesimen ini kemungkinan termasuk dalam genus yang berbeda dan telah diidentifikasi secara salah. Juga ditunjukkan oleh Chen et al. ( 2004 ) sebagai klade tunggal, UU1 (Konsensus Klade 16) didukung dengan suara bulat sebagai MOTU tunggal, meskipun mengandung empat genera yang berbeda ( Aiolomorphus rhopaloides Walker, Bephratelloides cubensis Ashmead, Phylloxeroxenus phylloxerae Ashmead dan Tenuipetiolus medicaginis Gahan). Selain itu, polifili dari genera Eurytoma dan Bruchophagus terbukti, menghasilkan beberapa spesimen singleton yang belum terselesaikan yang muncul di seluruh filogeni.
Klade 1–6 tampaknya termasuk dalam genus Tetramesa , sementara spesimen tak teridentifikasi lainnya dalam filogeni yang tersisa kemungkinan besar termasuk dalam genus eurytomid yang berbeda (Gambar 4 ). Klade 9 (NPNS.MIX.3) khususnya berisi spesimen yang ditetapkan sebagai morfospesies ‘ Tetramesa sp. 2’ dan ‘ Tetramesa sp. 5’, tetapi kemungkinan besar tidak benar.
Analisis jam molekuler
Filogeni Bayesian 28S rRNA yang diberi tanggal menempatkan asal-usul Eurytominae dalam kelompok antara Jura Tengah dan Kapur Bawah, pada perkiraan tanggal median 139,5 juta tahun lalu (Node B; 95% kepadatan posterior tertinggi [HPD] = 110,1–172,6 juta tahun lalu, pp = 1) (untuk semua referensi pada perkiraan tanggal node, lihat Gambar 5 dan Tabel 1 ). Ada dukungan bagi Eurytomidae sebagai kelompok yang jauh lebih muda relatif terhadap keluarga kelompok luar yang terdiri dari Chalcididae, Torymidae dan Megastigmidae (Node A; usia median = 287,7 juta tahun lalu, 95% HPD = 227,7–356,4 juta tahun lalu) dan subfamili Rileyinae.

TABEL 1. Estimasi waktu divergensi dalam jutaan tahun (usia median) untuk node yang ditunjukkan pada Gambar 5 , dengan interval kepercayaan (CI) kepadatan posterior tertinggi (HPD) 95%.
| simpul | Usia rata-rata | 95% HPD CI | simpul | Usia rata-rata | 95% HPD CI |
|---|---|---|---|---|---|
| A | 287.7 | 227,7–356,4 | H | 34.3 | 24,6–46 |
| B | 139.5 | 110.1–172.6 | SAYA | 18.3 | 7.3–31.5 |
| C | 128.5 | 102.1–158.9 | J | 11.5 | 4–21.2 |
| D | 104.4 | 78,9–134,4 | Bahasa Inggris: K | 19.7 | 16–27.4 |
| Bahasa Inggris | 67.1 | 47,4–91,8 | Saya | 31.9 | 28,8–37,4 |
| F | 50.2 | 36,3–67,9 | M | 19 | 9.3–30.7 |
| G | 44.5 | 31,9–59,6 | N | 23.8 | 22,9–28,5 |
Asal usul klade Tetramesa ditempatkan di antara Kapur Atas dan Eosen, pada usia rata-rata 67,1 juta tahun lalu (Node E; 95% HPD = 47,4–91,8, pp = 1). Klade PNS dan NPNS menyimpang sekitar 50,2 juta tahun lalu (Node F; 95% HPD = 36,3–67,9, pp = 0,95), dan ada beberapa bukti untuk potensi divergensi antara klade PNS NH dan Belahan Bumi Selatan (SH) sekitar 44,5 juta tahun lalu (Node G; 95% HPD = 31,9–59,6), meskipun nilai dukungan untuk node ini rendah (pp = 0,55), dan ada spesimen representatif yang terbatas untuk NH Tetramesa .
Klade Afrika Selatan mulai menyebar sekitar 34,3 juta tahun lalu (Node H; 95% HPD = 24,6–46, pp = 0,98), dengan diversifikasi cepat terjadi sekitar 5–15 juta tahun lalu (lihat plot garis keturunan melalui waktu yang disisipkan pada Gambar 5 ). NH T. romana dan T. bambusae tampaknya menyebar selama kerangka waktu relatif yang sama dengan kelompok Afrika Selatan (∼11,5 juta tahun lalu, Node H, pp = 1).
Spesimen Tetramesa yang tidak teridentifikasi yang dikumpulkan di Maryland, AS (JN623677), merupakan spesimen pertama yang terpisah dari klade utama sekitar 67 juta tahun lalu (Node E). Informasi lebih lanjut tentang spesimen ini tidak tersedia (tanaman inang, daerah asal), tetapi tampaknya spesimen ini mewakili garis keturunan unik yang mungkin terdiri dari kelompok yang lebih basal.
DISKUSI
Mendefinisikan apa yang merupakan ‘spesies’, dan bagaimana organisme hidup dapat digambarkan secara konsisten dan bermakna secara biologis, adalah topik yang telah lama diperdebatkan dalam biologi yang sedang berlangsung (De Queiroz 2005 ; Hey 2006 ; Wang, He, et al. 2020 ; Zachos 2018 ). Ada lebih dari 30 konsep spesies yang berbeda (Zachos 2016 ), dengan penambahan baru yang muncul secara teratur dalam literatur (Hill 2017 ; Hong 2020 ; Seifert 2020 ; Shanker et al. 2017 ). Salah satu tantangan utama untuk delimitasi spesies adalah membedakan antara divergensi tingkat spesies dan populasi (Derkarabetian et al. 2019 ). Jumlah spesies dapat ditaksir terlalu tinggi atau terlalu rendah karena sejumlah faktor, termasuk sensitivitas algoritma delimitasi terhadap struktur genetik populasi (misalnya, karena substruktur geografis) (Sukumaran & Knowles 2017 ), variasi ukuran sampel, pilihan penanda genetik, dan pelanggaran satu atau lebih asumsi statistik model (Carstens et al. 2013 ). Identifikasi taksonomi yang benar sangat penting dalam pengendalian biologis dan biologi invasi, karena pengendalian yang tidak efektif dan/atau invasi yang tidak terdeteksi dapat mengakibatkan kerusakan ekologi yang signifikan dan pemborosan sumber daya.
Analisis filogenetik dan pembatasan spesies saat ini mendukung keberadaan setidaknya 16 taksa SH Tetramesa yang baru bagi sains. Taksa ini menggunakan E. curvula , E. biflora , Eragrostis trichophora , Eragrostis plana , E. rigidior , S. pyramidalis , S. africanus , H. hirta dan A. gayanus sebagai inang rumput. Dukungan untuk klade PNS Tetramesa yang terdiri dari spesimen yang dikumpulkan pada E. trichophora selanjutnya didukung oleh morfologi empedu unik yang diamati di lapangan (van Steenderen et al. 2023 ). Hasil kami juga mendukung proposisi bahwa famili Eurytomidae adalah kelompok taksonomi yang relatif muda (Zerova 1992 ) yang mengalami radiasi cepat selama Miosen dan sesuai dengan Chen et al. ( 2004 ) dalam menyoroti (1) inkonsistensi dengan klasifikasi taksonomi eurytomid saat ini, dan khususnya Tetramesa ; (2) insiden tinggi klade yang belum terselesaikan dan polifiletik yang diamati pada pohon gen COI nuklir 28S dan mitokondria; dan (3) divergensi sekuens yang sangat bervariasi yang diamati, khususnya dalam data COI. Famili Eurytomidae diketahui polifiletik dari garis bukti molekuler dan morfologi (Campbell et al. 2000 ; Chen et al. 2004 ; Lotfalizadeh et al. 2007 ), dan, dengan demikian, saran revisi taksonomi kelompok tersebut dibenarkan (Gahan 1922 ).
Jumlah singleton yang ada dalam data COI mitokondria menghasilkan sejumlah besar estimasi MOTU dalam beberapa metode delimitasi, khususnya dalam keluaran ASAP dan bPTP. Dalam kasus metode berbasis pohon, hal ini kemungkinan besar terutama disebabkan oleh sifat filogeni COI yang belum terselesaikan dan prevalensi klade polifiletik (Kapli et al. 2017 ). Metode mPTP adalah yang paling konservatif baik dalam wilayah gen COI maupun 28S nuklir, terkadang memperkirakan kurang dari setengah jumlah MOTU yang diperkirakan oleh metode lain. Variasi dalam keluaran ini menyoroti pentingnya memasukkan beberapa uji delimitasi dalam studi taksonomi, karena pendekatan berbasis jarak dan berbasis pohon memiliki kelebihan dan peringatannya sendiri.
Metode pembatasan spesies harus didekati dengan pemahaman bahwa metode tersebut merupakan representasi yang tidak sempurna dari sistem biologis di dunia nyata (Carstens et al. 2013 ). Organisme aseksual (Dudgeon et al. 2017 ), populasi yang terfragmentasi secara alami, serta hibridisasi dan introgresi (Dowling & Secor 1997 ; Winter et al. 2018 ), misalnya, dapat menciptakan area abu-abu dalam batas spesies (Galtier 2019 ; Wiens 2007 ). Pendekatan terpadu direkomendasikan, menggunakan berbagai metode yang mencakup informasi genetik, biogeografi, perilaku, riwayat hidup, dan morfologi (disebut sebagai ‘taksonomi integratif’) (lihat, misalnya, Carstens et al. 2013 ; Hey 2006 ; Hey et al. 2003 ; Padial et al. 2010 ; Schlick-Steiner et al. 2010 ; Stanton et al. 2019 ; Yeates et al. 2011 ; Zamani et al. 2020 ). Langkah berikutnya dalam pekerjaan saat ini adalah memperoleh identifikasi morfologi dari ahli taksonomi untuk melengkapi data genetik.
Klade yang belum terselesaikan, seperti yang dilaporkan di sini, bukanlah hal yang jarang terjadi dalam filogeni hymenoptera dan telah dilaporkan dalam sejumlah famili lain, termasuk vespid ( Polistes Latreille) (Perrard et al. 2013 ; Santos et al. 2015 ), colletid ( Colletes Latreille) (Ferrari et al. 2020 ), agrid (Malagón-Aldana et al. 2022 ) dan ichneumonid (Gauld & Dubois 2006 ). Politomi biasanya menunjukkan data yang tidak mencukupi (‘politomi lunak’) tetapi, dalam beberapa kasus, menandakan peristiwa spesiasi simultan yang cepat (‘politomi keras’) (Walsh et al. 1999 ). Politom keras sering kali merupakan hasil dari isolasi simultan beberapa populasi karena peristiwa vikariansi (misalnya, glasiasi, kenaikan permukaan laut, dan perubahan lingkungan skala besar), yang mengarah pada spesiasi alopatrik atau peripatrik (Hoelzer & Meinick 1994 ). Radiasi cepat cichlid di Danau Malawi adalah contoh spesiasi politom yang terdokumentasi dengan baik, di mana diperkirakan bahwa ratusan spesies muncul dalam 2 juta tahun terakhir dari retakan Pliosen berikutnya yang membentuk Danau Malawi dan pegunungan Kipengere (Livingstone) (Genner et al. 2015 ; Scherz et al. 2022 ).
Meskipun ada peringatan yang terkait dengan penanggalan filogenetik yang hanya menggunakan laju jam molekuler (Emerson 2007 ; Pulquerio & Nichols 2007 ; Tiley et al. 2020 ), analisis penanggalan wilayah gen 28S rDNA dalam studi ini mendukung radiasi terkini Eurytomidae, dengan klade Tetramesa yang berasal antara Kapur Bawah dan Eosen dan mengalami laju diversifikasi yang cepat dalam 5–15 juta tahun terakhir. Radiasi cepat ini kemungkinan menjelaskan klade yang secara konsisten tidak terselesaikan dalam filogeni nuklir dan mitokondria dalam Chen et al. ( 2004 ), yang menunjukkan kemungkinan kasus politomi ‘keras’ sejati dalam genera eurytomid. Namun, lokus tambahan diperlukan untuk lebih jauh mendukung hipotesis ini. Tujuan dari setiap analisis penanggalan bukanlah untuk memperoleh tanggal absolut tetapi untuk membantu menjelaskan pola evolusi yang lebih luas. Eurytomidae telah dikenal karena tingkat diferensiasinya yang cepat ke dalam relung ekologi baru, yang mengarah pada spesialisasi inang (Zerova 1992 ). Selain itu, karena eurytomida fitofag dikaitkan secara eksklusif dengan inang tumbuhan angiosperma, diferensiasi ini telah dianggap menyertai diversifikasi tumbuhan angiosperma di SH (Gondwana) selama Kapur Bawah (145–100,5 juta tahun lalu) berdasarkan bukti fosil (Beaulieu et al. 2013 ; van der Kooi & Ollerton 2020 ; Zerova 1992 ).
Perkiraan rentang waktu asal usul Eurytominae dan Tetramesa bertepatan dengan radiasi Cretaceous pada angiospermae (Li et al. 2019 ; Zerova 1992 ) dan radiasi chalcidoid selama Eosen (Heraty & Darling 2009 ). Pola radiasi Miosen cepat yang terlihat pada kelompok PNS Tetramesa juga telah dilaporkan pada serangga lain (Aduse-Poku et al. 2022 ; Perrichot et al. 2022 ; Toussaint et al. 2012 ; Trewick & Morgan-Richards 2005 ; Winkler et al. 2009 ) dan bertepatan dengan perluasan padang rumput dan pencabutan habitat hutan di Afrika (Jacobs 2004 ; Osborne 2008 ; Uno et al. 2016 ). Perluasan padang rumput C4 selama Neogen dihipotesiskan didorong oleh pendinginan global, peningkatan kekeringan, penurunan CO2 atmosfer dan pola musim yang kuat yang menghasilkan lingkungan yang rentan terhadap kebakaran musiman (Beerling & Osborne 2006 ; Staver et al. 2011 ). Selain itu, telah disarankan bahwa fluktuasi iklim yang tiba-tiba selama Miosen memecah populasi dan mendorong diversifikasi garis keturunan (Zachos et al. 2008 ).
Ada dukungan untuk perpecahan antara NPNS dan PNS Tetramesa ~50,2 juta tahun lalu. Meskipun simpulnya tidak didukung dengan kuat (pp = 0,55), NH dan SH Tetramesa mungkin telah menyimpang ∼45 juta tahun lalu, dengan kelompok SH mulai terdiversifikasi ∼35 juta tahun lalu. Saat ini tidak mungkin untuk memastikan apakah Tetramesa berasal dari NH atau SH karena kurangnya data sekuens NH yang dikurasi secara menyeluruh dan bukti fosil yang dapat diandalkan. Namun, kerangka waktu ini cukup besar untuk menunjukkan kemungkinan perpecahan tingkat genus (Menezes et al. 2020 ; Samacá-Sáenz et al. 2022 ). Taksa yang telah menyebar baru-baru ini menimbulkan tantangan bagi metode pembatasan spesies karena mereka mungkin belum mencapai keadaan isolasi reproduktif atau monofili (Shaffer & Thomson 2007 ). Radiasi cepat cukup umum terjadi pada Hymenoptera parasitoid, yang diyakini terjadi karena pergantian inang untuk mengakses ceruk ekologi baru (Endara et al. 2018 ; Klopfstein et al. 2019 ; Peters et al. 2018 ; Smith et al. 2008 ). Hal ini tampaknya juga terjadi pada Tetramesa herbivora .
Karena sangat sedikit yang diketahui tentang pola pewarisan dan dinamika mitokondria dalam Eurytomidae (Chen et al. 2004 ), disarankan agar jam nuklir tambahan diselidiki untuk kelompok ini. Meskipun laju jam nuklir tunggal yang digunakan dalam penelitian ini (Papadopoulou et al. 2009 ) mungkin hanya memberikan perkiraan kasar waktu divergensi, hasil yang disajikan di sini masuk akal dalam konteks apa yang sudah diketahui dari catatan fosil dan peristiwa biogeografis historis. Sejumlah penelitian lain telah menggunakan jam nuklir untuk divergensi tingkat dalam dan terkini (Bertone et al., 2008 ; Kergoat et al., 2018 ; Meredith et al., 2008 ; Reidenbach et al., 2009 ; Schmitz et al., 2007 ; Steppan et al., 2005 ; Vea & Grimaldi 2016 ; Wiegmann et al., 2000 ), yang menunjukkan bahwa penanda nuklir terkadang lebih berguna dan kuat daripada gen mitokondria (Sato et al., 2016 ; Steppan et al., 2005 ) dan menawarkan wawasan berharga ke dalam pola diversifikasi.
Analisis genetik yang disajikan di sini telah mulai mengisi beberapa kesenjangan pengetahuan dalam taksonomi genus Tetramesa dan saat ini mencakup investigasi filogenetik paling komprehensif dari kelompok tersebut hingga saat ini. Pekerjaan ini telah menggabungkan penggunaan tanaman inang dan serangkaian metode pembatasan spesies untuk memperkirakan batas spesies dan menggabungkan laju jam molekuler nuklir untuk memperkirakan peristiwa divergensi historis. Temuan ini dapat berguna dalam klasifikasi taksonomi masa depan dan deskripsi spesies untuk kelompok tersebut, pemahaman tentang interaksi dan spesialisasi rumput-herbivora, pemilihan agen potensial untuk program pengendalian hayati rumput dan pemahaman keseluruhan tentang keanekaragaman mikrohimenoptera di SH.
